이번 포스팅에서는 영상 데이터 처리 방법에 대해 알아보도록 하겠습니다.
AI 모델의 성능을 높이기 위해서는 AI가 잘 학습할 수 있도록 입력 데이터를 잘 넣어주는 것이 중요합니다. 그러나 실제 현실의 데이터는 다양한 환경에서 수집될 수 있기 때문에 포맷이 제각각인 경우가 많은데요, 이를 해결하기 위해 반드시 거쳐야하는 과정이 preprocessing, 즉 데이터 전처리입니다.
그 중에서도 대표적으로 사용되는 기법인 resize, resample, normalization, gamma correction, adaptive equalization을 의료 영상 데이터를 활용하여 실습해보겠습니다!
데이터 및 라이브러리 가져오기
데이터를 현재 세션으로 가져옵니다.
!git clone https://github.com/Pulsar-kkaturi/DL-Education.git
일반적으로 의료 영상 데이터 처리에 사용되는 파이썬 라이브러리들을 모두 불러와봅시다.
코랩 환경 기준, simpleitk는 설치되어 있지 않기 때문에 별도로 설치해야해요.
# 의료영상 처리를 위한 라이브러리 설치
!pip install simpleitkimport numpy as np # 수치연산, 수학 라이브러리
import os # 파일 관리
import matplotlib # 수학적인 연산, 그래프 plottting
from matplotlib import pyplot as plt
from matplotlib.ticker import MultipleLocator
import SimpleITK as sitk # 의료영상용 라이브러리
import json
import csv
import pandas as pd
from sklearn import metrics as skmet
from skimage import morphology # 일반적인 영상처리
from skimage import measure
from skimage import exposure
from skimage.transform import rotate
from skimage import io as sio
from skimage import color as skc
import cv2 as cv
from keras.preprocessing import image as kimg데이터 확인
DICOM 포맷(.dcm) 데이터를 읽어들이기 위해서는 simpleitk 라이브러리가 필요합니다.
simpleitk의 ReadImage 메서드로 한번 데이터를 읽어봅시다.
test_data = './DL-Education/dataset/test.dcm'
image = sitk.ReadImage(test_data)
image
오잉 객체가 튀어나오는군요. 배열 형태로 바꿔줘야하나 봅니다.
GetArrayFromImage 메서드를 활용해서 배열 형태로 바꿔줍시다.
test_data = './DL-Education/dataset/test.dcm'
image = sitk.ReadImage(test_data)
img_arr = sitk.GetArrayFromImage(image)
print(img_arr.shape)
print(img_arr)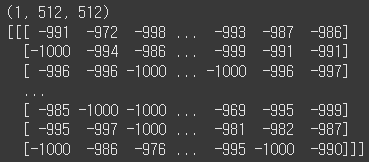
크기가 512x512인 배열이 나오는군요.
이제 simpleitk 라이브러리의 메서드들을 이용해서 데이터의 전반적인 정보를 살펴봅시다.
이미지 데이터니까 matplotlib를 이용해서 시각화도 한번 해보죠!
print('# Header Information #')
print('Image Size = ', image.GetSize()) # 이미지 크기
print('Pixel Spacing = ', image.GetSpacing()) # 픽셀 거리
print('Image Dimension = ', image.GetDimension()) # 원래 지축(높이)까지해서 3차원
print('Number of Pixel Components = ', image.GetNumberOfComponentsPerPixel()) # 흑백이라 1
print('Minimum & Maximum pixel value(Min/Max) = {}/{}'.format(np.min(img_arr), np.max(img_arr)))
print('Image mean & std = {}, {}'.format(np.mean(img_arr), np.std(img_arr)))
plt.figure(figsize=(5,5))
plt.imshow(img_arr[0], cmap='gray')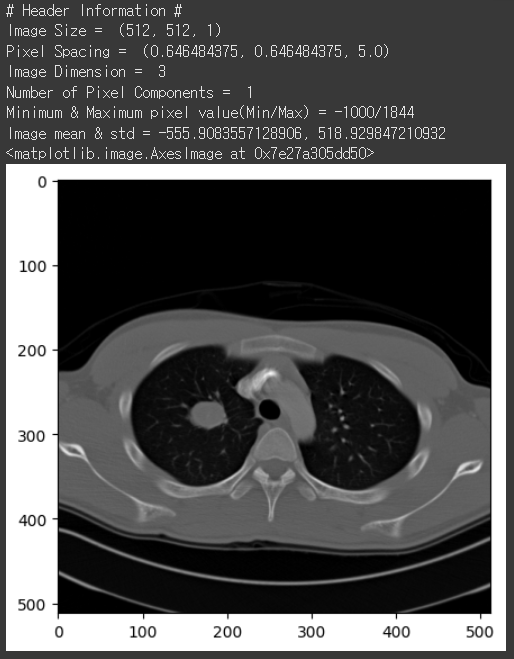
1. Image Size : 이미지의 크기(픽셀 수)
- 예 : (512, 512, 1)
- 가로 : 512 픽셀
- 세로 : 512 픽셀
- 깊이(Z축, 슬라이스 개수) : 1
2. Pixel Spacing : 각 픽셀 사이의 물리적 거리
- 예 : (0.646484375, 0.646484375, 5.0)
- 가로(X축) : 약 0.65mm
- 세로(Y축) : 약 0.65mm
- 깊이(Z축) : 5.0mm
3. Image Dimension : 이미지의 차원 수
- 예 : 3
- 3D 이미지(CT, MRI)
4. Number of Pixel Components : 한 픽셀당 데이터의 구성 요소 수 (일반적으로 흑백 or 컬러)
- 예 : 1
- 흑백 이미지(그레이 스케일)
5. Minimum & Maximum Pixel Value : 픽셀 값 중 최소값과 최대값 (이미지의 밝기 범위)
- 예 : -1000/1844
- 최소값 : -1000 (가장 어두운 픽셀)
- 최대값 : 1844 (가장 밝은 픽셀)
6. Image Mean & Std : 픽셀 값 평균과 표준편차 (이미지의 밝기 분포)
- 평균이 높다면 이미지가 전반적으로 밝은 상태
- 표준 편차가 크다면 밝기 차이가 크고, 작다면 고르게 분포
Resize
Resize는 말 그대로 이미지의 크기를 변화시키는 작업입니다.
Resize 함수를 만들어 모듈화 해보겠습니다.
# Resize
def resize_array(sitk_image, size, interpolator=sitk.sitkLinear):
original_spacing = sitk_image.GetSpacing()
original_size = sitk_image.GetSize()
new_size = list(original_size)
new_size[0]=size[0]
new_size[1]=size[1]
new_spacing = [(ospc * osz / nsz) for osz, ospc, nsz in
zip(original_size, original_spacing, new_size)]
sitk_image = sitk.Resample(sitk_image, new_size, sitk.Transform(), interpolator, sitk_image.GetOrigin(), new_spacing,
sitk_image.GetDirection(), 0, sitk_image.GetPixelID())
return sitk_image
원래 이미지는 512x512 였으니, 위 함수를 사용해서 256x256 크기로 줄여보죠.
resize_img = resize_array(image, [256, 256])
print('Original Image Size = ', image.GetSize())
print('Processed Image Size = ', resize_img.GetSize())
print('Original Pixel Spacing = ', image.GetSpacing())
print('Processed Pixel Spacing = ', resize_img.GetSpacing())
plt.figure(figsize=(12,5))
plt.subplot(1,2,1)
plt.imshow(img_arr[0], cmap='gray')
plt.subplot(1,2,2)
plt.imshow(sitk.GetArrayFromImage(resize_img)[0], cmap='gray')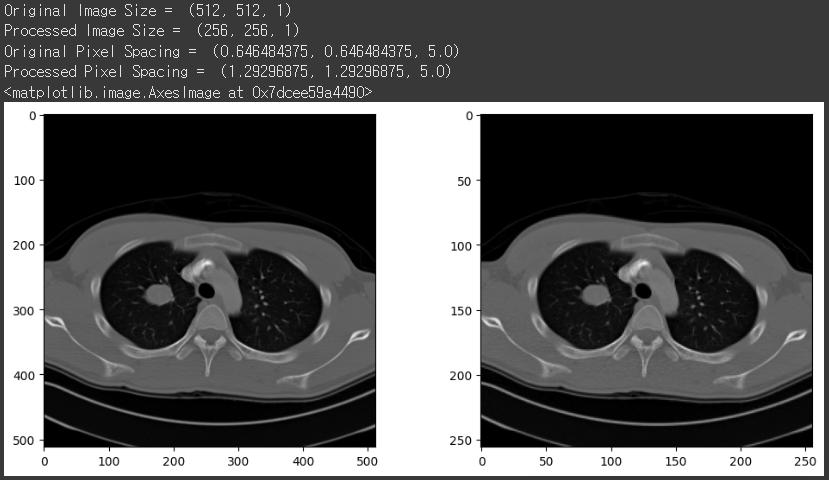
같은 이미지에서 픽셀의 개수를 줄이면 당연히 한 픽셀이 커버하는 실제 세계에서의 크기(pixel spacing)는 커지게 되겠죠?
512x512에서 256x256으로 크기를 줄였더니, pixel spacing이 커진 것을 통해 이 사실을 확인할 수 있었습니다!
Resample
Resample은 픽셀 간 물리적 거리를 변화시키는 작업입니다.
Resample 함수를 만들어 모듈화 해보겠습니다.
# Resample
def resample_array(sitk_image, spacing, interpolator=sitk.sitkLinear):
original_spacing = sitk_image.GetSpacing()
original_size = sitk_image.GetSize()
new_spacing = [spacing, spacing, original_spacing[2]]
new_size = [int(round(osz * ospc / nspc)) for osz, ospc, nspc in
zip(original_size, original_spacing, new_spacing)]
sitk_image = sitk.Resample(sitk_image, new_size, sitk.Transform(), interpolator, sitk_image.GetOrigin(), new_spacing,
sitk_image.GetDirection(), 0, sitk_image.GetPixelID())
return sitk_image
원래 픽셀 간 거리가 (0.646484375, 0.646484375, 5.0)로 딱 떨어지지 않는 숫자였습니다.
위 함수를 사용해서 (1.0, 1.0, 5.0)으로 픽셀 간 거리를 늘려보겠습니다.
resample_img = resample_array(image, 1)
print('Original Pixel Spacing = ', image.GetSpacing())
print('Processed Pixel Spacing = ', resample_img.GetSpacing())
print('Original Image Size = ', image.GetSize())
print('Processed Image Size = ', resample_img.GetSize())
plt.figure(figsize=(12,5))
plt.subplot(1,2,1)
plt.imshow(img_arr[0], cmap='gray')
plt.subplot(1,2,2)
plt.imshow(sitk.GetArrayFromImage(resample_img)[0], cmap='gray')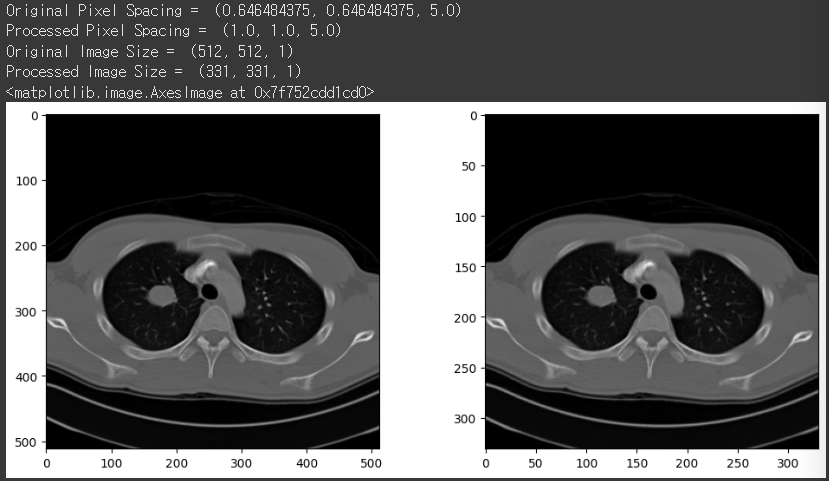
한 픽셀이 커버하는 현실 세계에서의 범위가 커지면, 당연히 픽셀의 개수는 줄어들게 되겠죠?
pixel spacing을 줄였더니, 이미지 크기(픽셀 개수)가 작아진 것을 통해 이 사실을 확인할 수 있었습니다!
Normalization
이미지 데이터는 픽셀 값이 다양한 범위를 가질 수 있습니다.
보통 일반적인 이미지는 0~255 사이의 값을 가지지만, CT나 MRI 같은 의료 영상은 -1000~4000까지 값을 가지기도 하죠.
이렇게 다양한 이미지들의 범위를 통일하지 않으면 모델을 학습할 때 특정 값 범위가 큰 데이터가 더 큰 영향을 미쳐 학습이 비효율적일 수 있습니다.
그래서 필요한 것이 바로 Normalization!
대표적으로 사용되는 Min-Max 정규화와 Z-score 정규화에 대해 알아보겠습니다.
Min-Max Normalization
Min-Max 정규화는 이미지의 픽셀 값들을 0과 1 사이의 값으로 바꾸어 정규화시키는 방법입니다.
# MinMax Normalization
norm_img = (img_arr - np.min(img_arr)) / (np.max(img_arr) - np.min(img_arr))
print('Oringinal Image min/max value = {}/{}'.format(np.min(img_arr), np.max(img_arr)))
print('Processed Image min/max value = {}/{}'.format(np.min(norm_img), np.max(norm_img)))
print('\nSample Patch Comparison(Origin vs Processed)')
print(img_arr[0, 275:280, 300:305])
print(norm_img[0, 275:280, 300:305])
plt.figure(figsize=(12,5))
plt.subplot(1,2,1)
plt.imshow(img_arr[0], cmap='gray')
plt.subplot(1,2,2)
plt.imshow(norm_img[0], cmap='gray')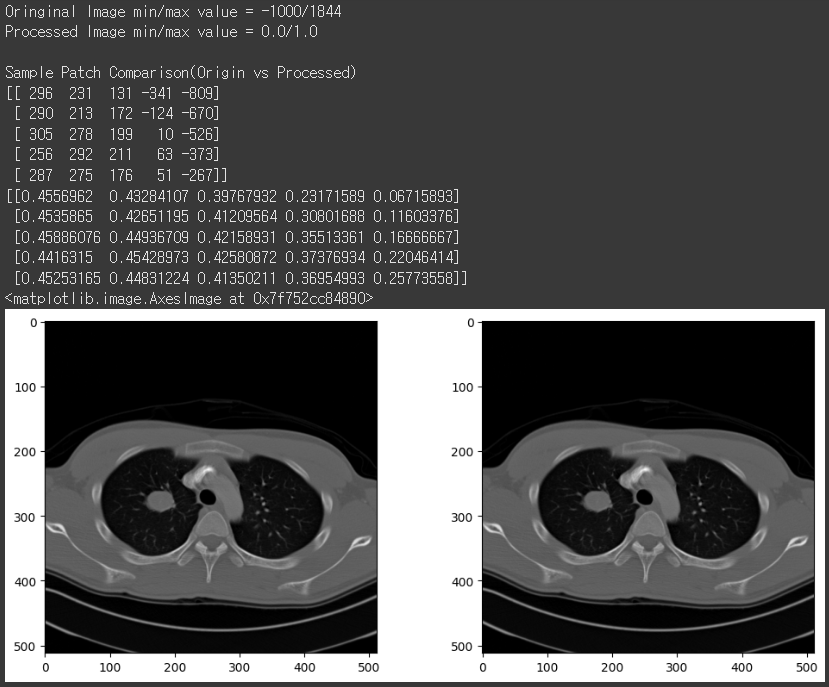
이미지에는 전혀 영향이 없지만, 수치를 보면 0과 1 사이로 바뀌어 있음을 확인할 수 있습니다.
Z-Score Normalization
Z-Score 정규화는 Z-score를 이용하여 이미지의 픽셀 값들이 정규분포를 따르도록 정규화시키는 방법입니다.
# Z-Score Normalization
zsc_img = (img_arr - np.mean(img_arr)) / np.std(img_arr)
print('Oringinal Image min/max value = {}/{}'.format(np.min(img_arr), np.max(img_arr)))
print('Processed Image min/max value = {}/{}'.format(np.min(zsc_img), np.max(zsc_img)))
print('Oringinal Image mean/std value = {}/{}'.format(np.mean(img_arr), np.std(img_arr)))
print('Processed Image mean/std value = {}/{}'.format(np.mean(zsc_img), np.std(zsc_img)))
print('\nSample Patch Comparison(Origin vs Processed)')
print(img_arr[0, 275:280, 300:305])
print(zsc_img[0, 275:280, 300:305])
plt.figure(figsize=(8,8))
plt.imshow(zsc_img[0], cmap='gray')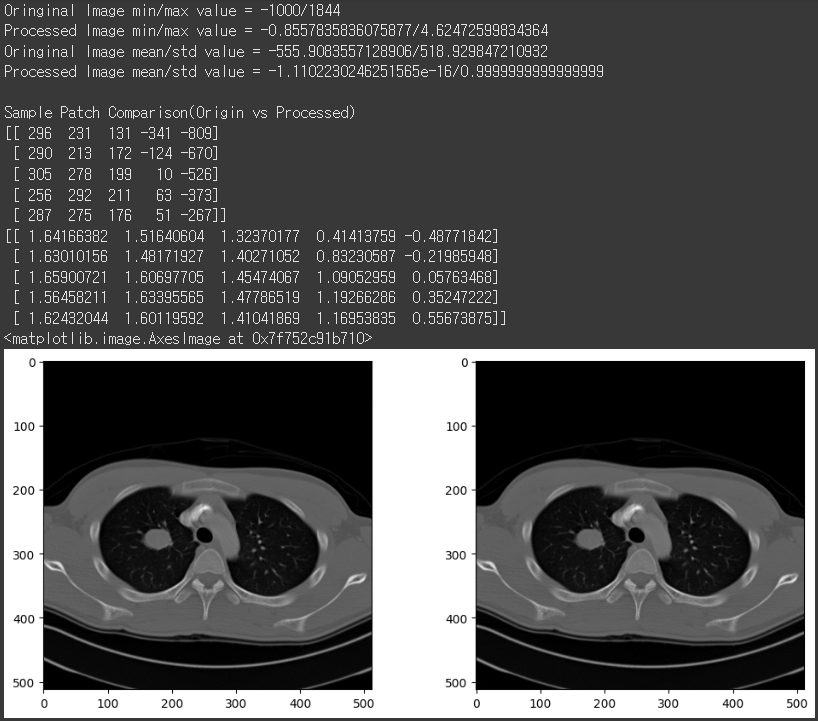
이 역시 이미지에는 전혀 영향이 없지만, 수치를 보면 표준화된 수치로 바뀌어 있음을 확인할 수 있습니다.
Gamma Correction
Gamma Correction은 이미지의 밝기(Luminance)를 조정하는 과정입니다.
이게 왜 필요할까요?
감마 보정을 하지 않으면 어두운 영역이 너무 어둡거나 밝은 영역이 너무 밝아져 디테일이 사라질 수 있습니다.
감마 보정을 하게되면, 어두운 영역과 밝은 영역을 적절히 보정해, 이미지 디테일을 더 잘 표현할 수 있게 되는 것이죠!
Gamma correction의 공식은 간단합니다. 그냥 감마만큼 제곱해주는 거에요.
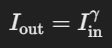
- γ > 1 : 어두운 부분을 강조(밝은 부분을 줄임) → 이미지가 더 어두워짐
- γ < 1 : 밝은 부분을 강조(어두운 부분을 줄임) → 이미지가 더 밝아짐
- γ = 1 : 원본 이미지와 동일

skimage 라이브러리의 exposure 모듈에서 adjust_gamma 메서드를 활용하면 쉽게 gamma correction을 수행할 수 있습니다!
여기서 중요한 것은 input이 uint8 타입인 경우 반드시 min-max 정규화 시킨 후에 255를 곱해서 gamma correction을 진행해야 한다는 것!!
☞ Scikit-Image 라이브러리는 uint8 타입 input에 대해 기본적으로 0~255 범위의 입력을 처리하도록 설계되어 있기 때문
만약 input이 uint8 타입이 아니고 float라면 min-max 정규화만 수행하여 0~1 범위로만 만들고 gamma correction을 진행하는게 맞습니다!
☞ 공식 scikit-image 코드 참조 : https://github.com/scikit-image/scikit-image/blob/v0.25.2/skimage/exposure/exposure.py#L626-L686
# Gamma Correction
gamma_cor = exposure.adjust_gamma(255*norm_img, 0.5)
print('Oringinal Image mean/min/max value = {}/{}'.format(np.mean(255*norm_img), np.std(255*norm_img)))
print('Processed Image mean/min/max value = {}/{}'.format(np.mean(gamma_cor), np.std(gamma_cor)))
plt.figure(figsize=(12,6))
plt.subplot(1,2,1)
plt.imshow(img_arr[0], cmap='gray')
plt.subplot(1,2,2)
plt.imshow(gamma_cor[0], cmap='gray')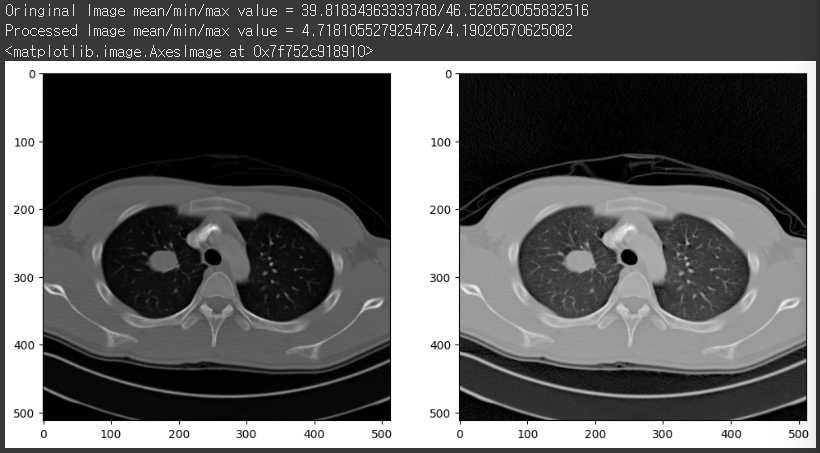
감마를 0.5로 했더니 이미지가 더 밝아진 것을 확인할 수 있습니다!
이러한 이미지들이 input으로 더 추가되면 모델 성능을 더 높일 수 있을 것 같네요~
Adaptive Equalization (적응형 히스토그램 균등화)
Adaptive Equalization은 이미지의 밝기와 대비(contrast)를 개선하기 위한 기법입니다.
이미지의 전체적인 밝기가 균일하지 않고, 어두운 영역과 밝은 영역이 동시에 존재하는 경우, 각 영역의 대비를 독립적으로 조정하여 더 많은 디테일을 드러나게 합니다.
skimage 라이브러리의 exposure 모듈에서 equalize_adapthist 메서드를 활용하면 쉽게 adaptive equalization을 수행할 수 있습니다!
# Adaptive Equalization
img_norm = (img_arr - np.min(img_arr))/(np.max(img_arr)-np.min(img_arr))
img_eqh = exposure.equalize_adapthist(img_norm, clip_limit=0.02)
plt.figure(figsize=(12,6))
plt.subplot(1,2,1)
plt.imshow(img_arr[0], cmap='gray')
plt.subplot(1,2,2)
plt.imshow(img_eqh[0], cmap='gray')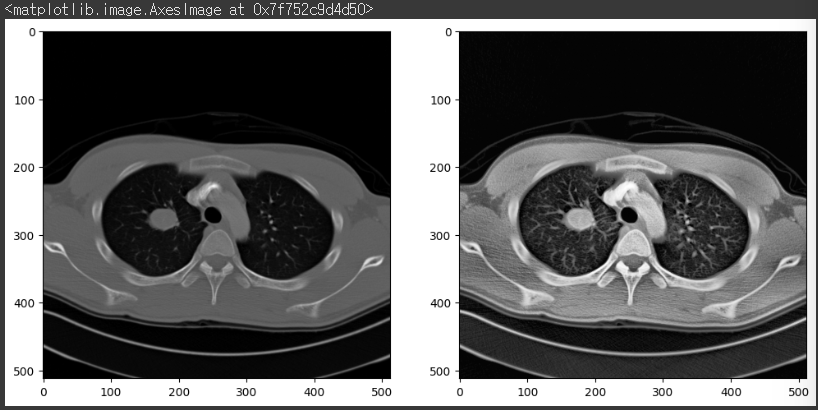
놀랍지 않나요?
이전까지 보이지 않았던 디테일들이 눈에 명확히 들어오는 것을 확인할 수 있습니다.
'Medical Imaging > Preprocess' 카테고리의 다른 글
| HD-CTBET 툴을 사용하여 CT에서 skull stripping하는 방법 (1) | 2025.06.04 |
|---|---|
| HD-BET 툴을 사용하여 MRI에서 skull stripping하는 방법 (1) | 2025.06.04 |
| SynthStrip 사용하여 skull stripping 하는 방법 (2) | 2025.06.04 |
| ANTsPy 라이브러리를 활용한 정합(registration) (1) | 2025.05.27 |



