U-Net은 의료 영상 도메인의 정밀한 픽셀 단위 분할(Segmentation)이 필요한 과제에서 가장 널리 사용되는 구조입니다.
저도 회사에서 brain의 stroke lesion을 segmentation하는 모델을 만드는 프로젝트에 참여하게 되었는데, 기본적으로 U-Net 구조를 베이스로 하는 모델을 튜닝하고 있기 때문에, 더 자세히 공부하고 기록하기 위해 포스팅을 쓰게 되었습니다.
이번 포스트에서는 U-Net의 구조에 대해 각 블록이 무엇을 의미하는지, 왜 그렇게 설계되었는지까지 한 블록씩 파헤쳐보겠습니다.
전체 구조 요약
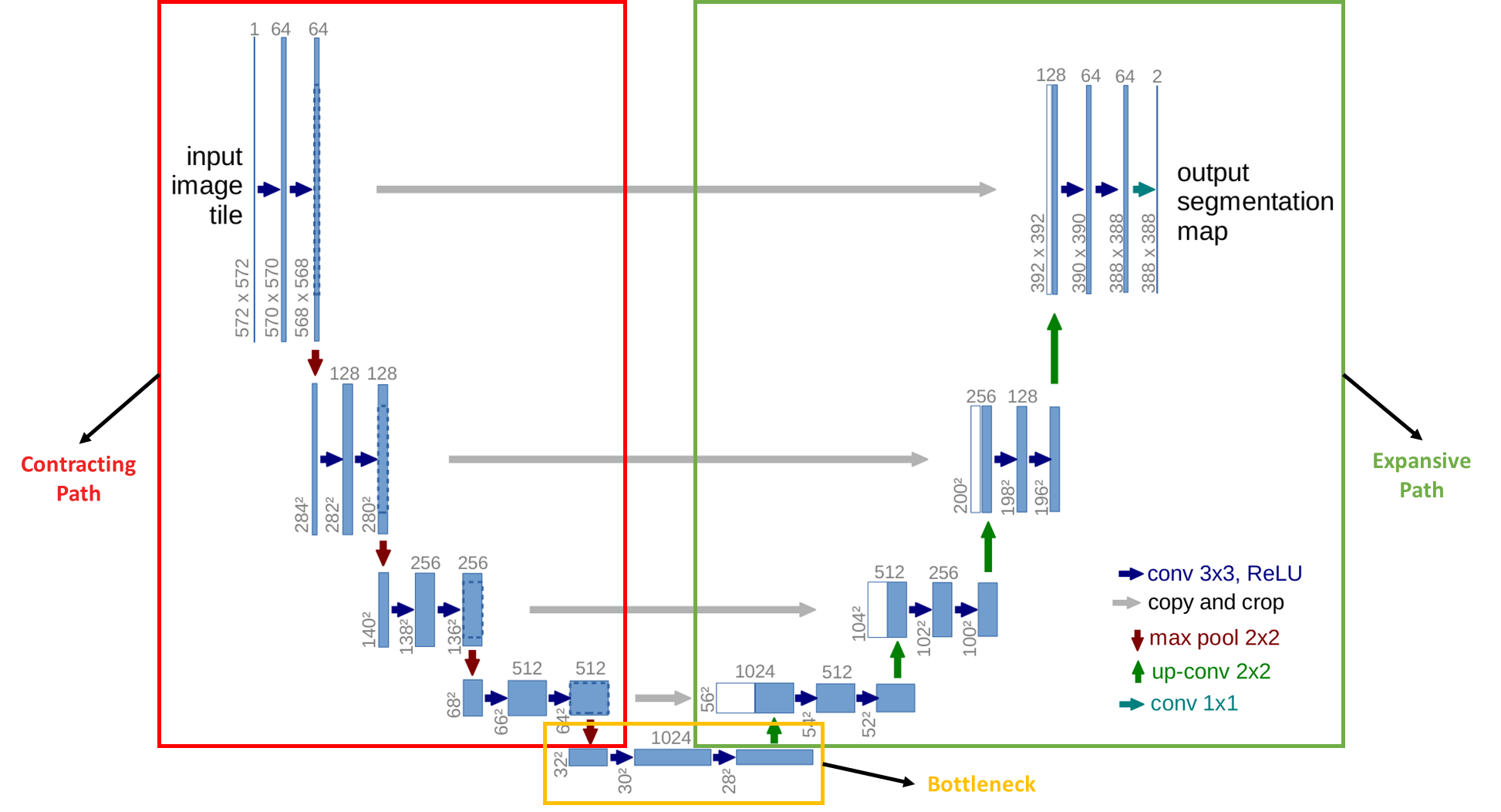
- Contracting Path
- 특징 추출(Feature extraction)
- Expansive Path
- 자세한 위치 복원(Spatial information 복구)
- Bottleneck
- Skip Connection
- "위치 정보" 를 잃지 않고 복구 가능하게 함
추가적으로, 위와는 다른 방식이지만 나름 이해하기 쉽게 잘 정리되어 있는 figure가 또 있길래 이것도 첨부합니다~

블록별 구성 분석
🔹 입력 & 첫 Conv 블록

1. input이 들어옵니다. (572×572×1)
2. Conv(3×3) → ReLU를 통과시킵니다. (570×570×64)
- 필터의 개수를 64개로 해서 결과도 64 채널이 나오게 됩니다.
- 572×572에서 570×570으로 변한 이유는 padding을 안했기 때문이죠.
3. 한 번 더 같은 조건으로 Conv(3×3) → ReLU를 통과시킵니다. (568×568×64)
- 이번에도 같은 조건이지만, 한가지 다른 점은 앞선 convolution 때문에 채널이 64개가 된 상태라, 각 필터의 채널도 64개로 맞춰 주어야 겠죠. 즉, 필터 하나의 shape이 (3×3×64)가 될 겁니다.
4. Max Pooling(2×2)을 수행합니다. (284×284×64)
- 2×2 필터로 pooling을 진행했으니 이미지 크기는 절반으로 줄어들겠죠!
- 당연히 채널 수에는 영향이 없습니다.
🔹 Contracting Path: Downsampling

1. Conv(3×3) → ReLU를 통과시킵니다. (282×282×128)
- 필터의 개수를 두배로 늘려 128개로 해서 결과도 128 채널이 나오게 됩니다.
- 패딩을 안했기 때문에 이미지 크기는 가로 세로 각각 2픽셀씩 줄어들게 됩니다.
2. 한 번 더 Conv(3×3) → ReLU를 통과시킵니다. (280×280×128)
3. Max Pooling(2×2)을 수행합니다. (140×140×128)
4. 1~3번까지의 과정을 2번 더 반복합니다. (32×32×512)
🔹 Bottleneck

1. Conv(3×3) → ReLU를 통과시킵니다. (32×32×512) → (30×30×1024)
- 앞선 convolution 방식과 완전히 동일하지만, 채널이 512에서 1024로 크게 늘어나면서 가장 깊은 표현이 이루어지게 됩니다.
2. 한 번 더 Conv(3×3) → ReLU를 통과시킵니다. (28×28×1024)
🔹 Expansive Path: Upsampling
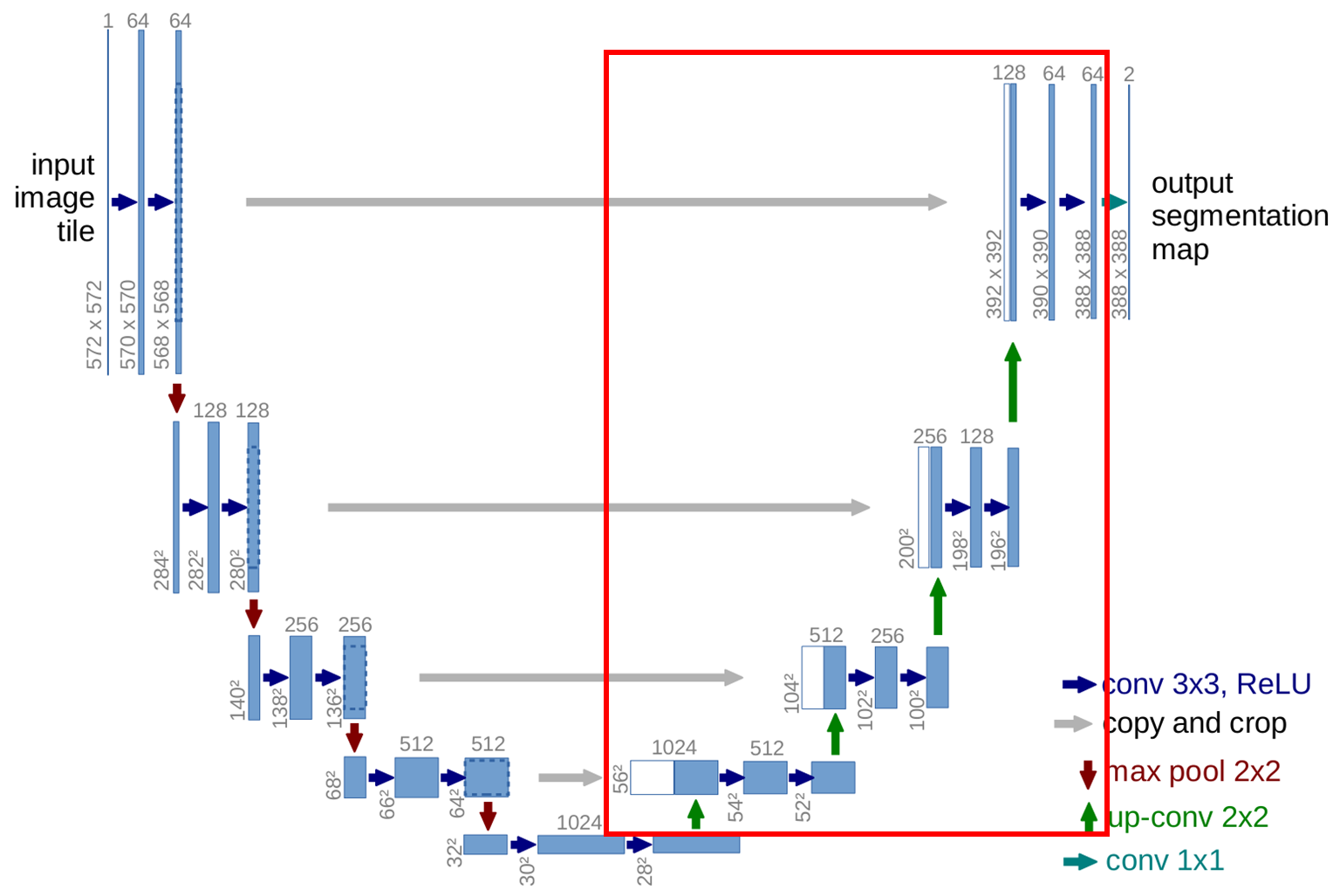
1. Up-Convolution(2×2)을 수행합니다. (stride=2) (28×28×1024) → (56×56×512)
- 2×2 필터로 stride=2 Up-convolution을 수행했기에 이미지 크기는 2배가 됩니다.
- 필터 개수는 절반으로 줄여 채널을 절반으로 줄어들게 만들었습니다.
2. 해상도가 줄면서 나타나는 위치정보 손실 방지를 위해 같은 단계의 encoder feature map을 복사(crop) 후 concatenate 해줍니다. (56×56×1024)
- encoder 부분은 64×64이고, decoder 부분은 56×56이므로 encoder 부분에서 가운데 56×56 부위를 crop합니다.
- concatenate은 단순 덧셈이 아닌 채널 방향으로 이어붙입니다. 즉, encoder의 512 채널과 decoder의 512 채널을 붙여서 1024 채널로 만드는 것이죠.
3. Conv(3×3) → ReLU를 통과시킵니다. (54×54×512)
- Conv를 통해 concatenate에 의해 다시 2배로 늘어났던 채널 개수를 절반으로 정상화 시켜줍니다.
4. 한 번 더 Conv(3×3) → ReLU를 통과시킵니다. (52×52×512)
5. 1~4번까지의 과정을 3번 더 반복합니다. (388×388×64)
🔹 출력층: 1×1 Convolution
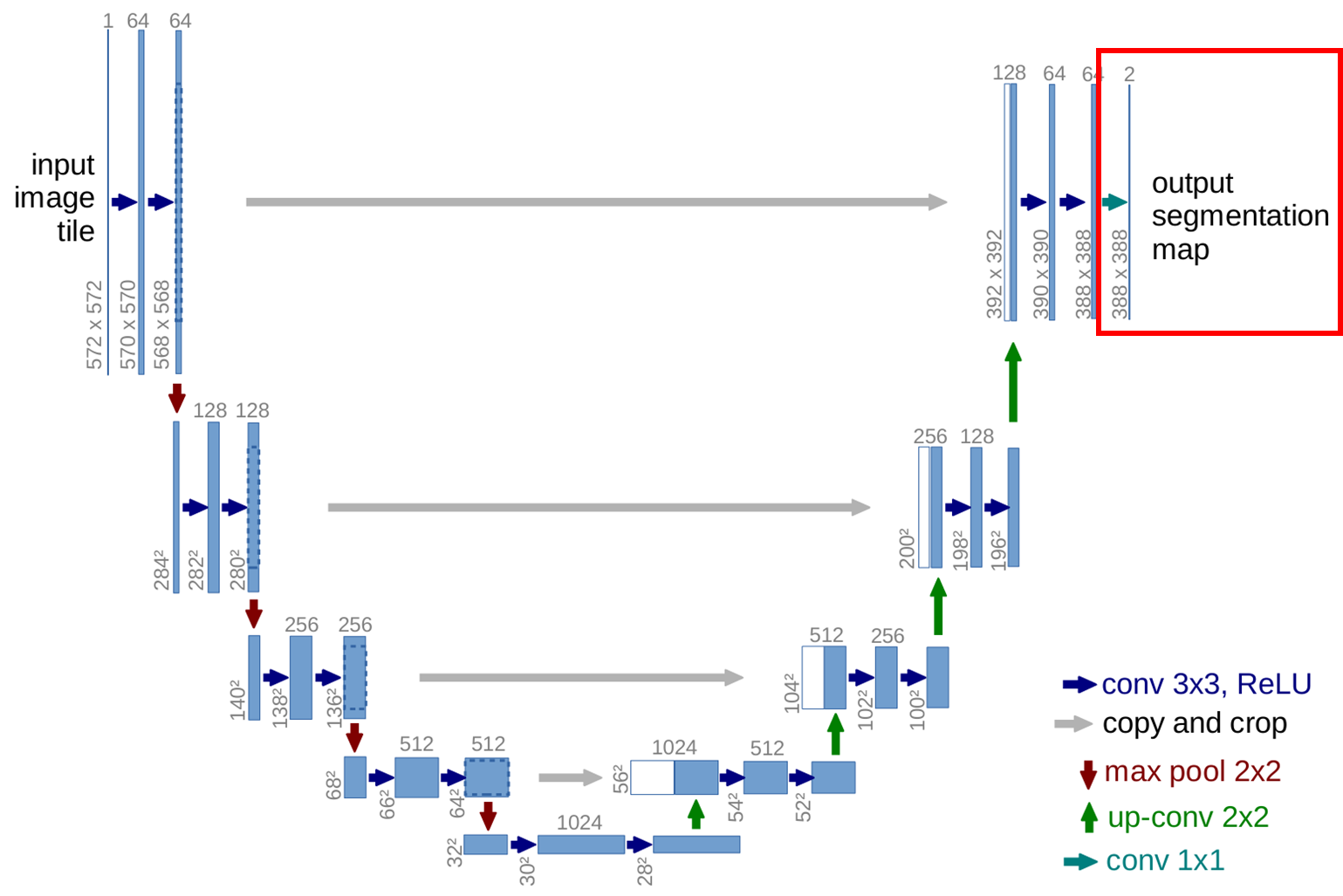
1. Conv(1×1) 통과시킵니다. (388×388×64) → (388×388×2)
- (1×1) 크기의 convolution 필터를 통해 공간 정보는 건드리지 않고 채널 간 정보만 결합합니다.
- 다시 말해, 1×1 Conv는 각 위치의 채널 벡터를 선형 결합해 클래스 확률 벡터를 생성해줍니다. (위 그림에서는 2개의 클래스 출력)
- 의료 도메인에서는 세포 vs 배경, 병변 vs 정상 등으로 분할이 가능할 것입니다.
지금은 softmax를 사용했기 때문에 최종 output의 채널이 2개인데, "병변 vs 정상"과 같이 클래스가 2개인 경우 binary 문제이므로 sigmoid를 사용해도 됩니다. sigmoid를 사용할 경우 output의 채널은 1개가 되겠죠.
'Study > AI' 카테고리의 다른 글
| [AI] Dice Coefficient와 IoU(Intersection over Union) 비교 (0) | 2025.04.29 |
|---|---|
| [AI] 전이학습(Transfer Learning)에 대하여 (0) | 2025.03.05 |
| [AI] K-Fold Cross Validation에 대하여 (0) | 2025.03.03 |
| [AI] 딥러닝 개발을 위한 CPU, GPU, 메모리 이해하기 (3) | 2025.02.07 |


